生物多様性指数の算出や群集構造解析(クラスター解析・NMDS)を行いたい場合、フリーソフトウェアで統計解析向けのプログラミング言語『R』を用いて行うのが最適でしょう。しかし、私が学生の頃は資料が少なく、コードを書くのに苦労したものです。変数の代入(定義)シンボルや引数の表現の揺れや関数のネーミングなどもサイトによって異なり、かなり不統一でした。今は表現の統一化が進み、ChatGPTといった優れた生成AIの登場によって、かなり読みやすい言語にはなったとは思いますが、改めてこの記事では私が行った土壌動物の同定データから群集構造解析を行った例をご紹介してみようと思います。
ある地点での土壌動物分析例
以下はある4地点、2シーズンのツルグレン法によって抽出された土壌動物サンプルの同定結果です。コウチュウ目を除く種を私が担当しました。このデータで生物多様性指数算出・群集構造解析を行っていきましょう。
以下は生データであるため、実際にはここから地点・調査日ごとの入力種名の個体数を集計したものを解析することになりますが、ここでは省略します。
| 入力種名 | 性別 | 個体数 | 調査日 | 調査地点 |
| ヨロイジュズダニ | 2 | 5月 | A | |
| ハナビラオニダニ | 2 | 5月 | A | |
| Xylobates属sp. | 21 | 5月 | A | |
| ヒラタオニダニ | 1 | 5月 | A | |
| アジアオニダニ | 1 | 5月 | A | |
| ヒワダニ | 20 | 5月 | A | |
| ワタゲジュズダニ | 3 | 5月 | A | |
| ジュズダニ科sp. | 8 | 5月 | A | |
| ヤマトクモスケダニ | 2 | 5月 | A | |
| クワガタダニ | 3 | 5月 | A | |
| ヒメヘソイレコダニ | 2 | 5月 | A | |
| オバケツキノワダニ | 1 | 5月 | A | |
| トウキョウフリソデダニ | 3 | 5月 | A | |
| Veigaia属sp. | 9 | 5月 | A | |
| Parasitus属sp. | 2 | 5月 | A | |
| イトダニ科sp. | 9 | 5月 | A | |
| Podocinum属sp. | 1 | 5月 | A | |
| Pachylaelaps属sp. | 1 | 5月 | A | |
| Epicrius属sp. | 6 | 5月 | A | |
| トゲダニ目spp. | 24 | 5月 | A | |
| シマツノアヤトビムシ | 1 | 5月 | A | |
| アイイロハゴロモトビムシ | 2 | 5月 | A | |
| コガタドウナガトビムシ | 7 | 5月 | A | |
| ニッポンシロトビムシ | 1 | 5月 | A | |
| フクロムラサキトビムシ | 3 | 5月 | A | |
| ヨシイフォルソムトビムシ | 1 | 5月 | A | |
| カマアシムシ目sp. | 2 | 5月 | A | |
| ニセハリアリ | 6 | 5月 | A | |
| ウメマツオオアリ | 1 | 5月 | A | |
| テラニシハリアリ | 1 | 5月 | A | |
| コムカデ綱 | 2 | 5月 | A | |
| タカミヒトフシムカデ | ♀ | 1 | 5月 | A |
| タジマガハラヒトフシムカデ | ♀ | 1 | 5月 | A |
| Monotarsobius属sp. | ♂ | 1 | 5月 | A |
| オウギヤスデ | 1 | 5月 | A | |
| ミコシヤスデ科sp. | 1 | 5月 | A | |
| フイリワラジムシ | 1 | 5月 | A | |
| ナガワラジムシ | 8 | 5月 | A | |
| イトダニ科sp. | 1 | 5月 | A | |
| ヒワダニ | 2 | 5月 | A | |
| ヒメミミズ科sp. | 10 | 5月 | A | |
| アイイロハゴロモトビムシ | 1 | 5月 | B | |
| シマツノアヤトビムシ | 3 | 5月 | B | |
| ヒメトゲトビムシ | 9 | 5月 | B | |
| フクロムラサキトビムシ | 15 | 5月 | B | |
| コツノアリ | 11 | 5月 | B | |
| ミズアブ科sp. | 1 | 5月 | B | |
| コムカデ綱 | 1 | 5月 | B | |
| ヒラタオニダニ | 1 | 5月 | B | |
| クワガタダニ | 1 | 5月 | B | |
| ワタゲジュズダニ | 2 | 5月 | B | |
| Podocinum属sp. | 1 | 5月 | B | |
| トゲダニ目spp. | 3 | 5月 | B | |
| アザミウマ目sp. | 1 | 5月 | B | |
| ヤブコウジハカマカイガラムシ | 1 | 5月 | B | |
| ワタナベヒトフシムカデ酷似種 | ♀♂ | 2 | 5月 | B |
| Monotarsobius属sp. | ♂ | 4 | 5月 | B |
| コムカデ綱 | 3 | 5月 | B | |
| ワタゲジュズダニ | 23 | 5月 | B | |
| ジュズダニ科sp. | 2 | 5月 | B | |
| クワガタダニ | 7 | 5月 | B | |
| ウスイロヒメヘソイレコダニ | 1 | 5月 | B | |
| Xylobates属sp. | 5 | 5月 | B | |
| オタフクダルマヒワダニ | 1 | 5月 | B | |
| Podocinum属sp. | 2 | 5月 | B | |
| Holaspina属sp. | 4 | 5月 | B | |
| Pachylaelaps属sp. | ♂ | 4 | 5月 | B |
| Parasitus属sp. | 1 | 5月 | B | |
| トゲダニ目spp. | 47 | 5月 | B | |
| Abrolophus属sp. | 1 | 5月 | B | |
| ハナビラオニダニ | 1 | 5月 | C | |
| ヨロイジュズダニ | 1 | 5月 | C | |
| ワタゲジュズダニ | 10 | 5月 | C | |
| Xylobates属sp. | 46 | 5月 | C | |
| ウスイロヒメヘソイレコダニ | 1 | 5月 | C | |
| アラメイレコダニ | 1 | 5月 | C | |
| ヤマトクモスケダニ | 3 | 5月 | C | |
| ヒワダニ | 2 | 5月 | C | |
| クチバシツブダニ | 1 | 5月 | C | |
| ツルトミイチモンジダニ | 1 | 5月 | C | |
| Podocinum属sp. | 3 | 5月 | C | |
| イトダニ科sp. | 5 | 5月 | C | |
| Veigaia属sp. | 1 | 5月 | C | |
| Holostaspella属sp. | 4 | 5月 | C | |
| Holaspina属sp. | 2 | 5月 | C | |
| トゲダニ目spp. | 12 | 5月 | C | |
| ケダニ目sp. | 1 | 5月 | C | |
| カマアシムシ目sp. | 2 | 5月 | C | |
| フクロムラサキトビムシ | 2 | 5月 | C | |
| アイイロハゴロモトビムシ | 4 | 5月 | C | |
| シマツノアヤトビムシ | 3 | 5月 | C | |
| キタウロコアリ | 1 | 5月 | C | |
| ニセハリアリ | 16 | 5月 | C | |
| テラニシハリアリ | 1 | 5月 | C | |
| コツノアリ | 26 | 5月 | C | |
| チョウ目sp. | 1 | 5月 | C | |
| フイリワラジムシ | 5 | 5月 | C | |
| ナガワラジムシ | 1 | 5月 | C | |
| ムサシアカムカデ | 1 | 5月 | C | |
| オウギヤスデ | 2 | 5月 | C | |
| ミコシヤスデ科sp. | 2 | 5月 | C | |
| Monotarsobius属sp. | ♂ | 6 | 5月 | C |
| サカヨリチビヒトフシムカデ酷似種 | ♂ | 1 | 5月 | C |
| Bothropolys属sp. | 1 | 5月 | C | |
| Pheretima属sp. | 1 | 5月 | C | |
| ヒメミミズ科sp. | 1 | 5月 | C | |
| フタツメフォルソムトビムシ | 3 | 5月 | D | |
| フクロムラサキトビムシ | 2 | 5月 | D | |
| ニッポンシロトビムシ | 3 | 5月 | D | |
| ツチカメムシ科sp. | 4 | 5月 | D | |
| ウロコアリ | 1 | 5月 | D | |
| コツノアリ | 29 | 5月 | D | |
| アメイロアリ | 1 | 5月 | D | |
| ミズアブ科sp. | 3 | 5月 | D | |
| イトダニ科sp. | 3 | 5月 | D | |
| イトダニ科sp. | 8 | 5月 | D | |
| Holostaspella属sp. | 4 | 5月 | D | |
| トゲダニ目spp. | 10 | 5月 | D | |
| Xylobates属sp. | 11 | 5月 | D | |
| Lasioseius属sp. | ♂♀ | 2 | 5月 | D |
| ミナミクモスケダニ | 6 | 5月 | D | |
| トウキョウフリソデダニ | 9 | 5月 | D | |
| ワタゲジュズダニ | 1 | 5月 | D | |
| ヒメヘソイレコダニ | 1 | 5月 | D | |
| ヒメイブリダニ | 1 | 5月 | D | |
| コムカデ綱 | 1 | 5月 | D | |
| ミコシヤスデ科sp. | 9 | 5月 | D | |
| ゴシチナガズムカデ | 1 | 5月 | D | |
| Monotarsobius属sp. | 1 | 5月 | D | |
| フイリワラジムシ | 1 | 5月 | D | |
| ナガワラジムシ | 9 | 5月 | D | |
| Pheretima属sp. | 1 | 5月 | D | |
| ヒメミミズ科sp. | 1 | 5月 | D | |
| コツノアリ | 1 | 8月 | A | |
| ヤブコウジハカマカイガラムシ. | 3 | 8月 | A | |
| Mecistocephalus属sp. | 1 | 8月 | A | |
| Prolamnonyx属sp. | 1 | 8月 | A | |
| フイリワラジムシ | 3 | 8月 | A | |
| ミナミクモスケダニ | 3 | 8月 | A | |
| ハナビラオニダニ | 1 | 8月 | A | |
| アジアオニダニ | 2 | 8月 | A | |
| ナミツブダニ | 1 | 8月 | A | |
| オバケツキノワダニ | 1 | 8月 | A | |
| クワガタダニ | 2 | 8月 | A | |
| ツルトミイチモンジダニ | 1 | 8月 | A | |
| ヒワダニ | 1 | 8月 | A | |
| ワタゲジュズダニ | 1 | 8月 | A | |
| ジュズダニ科sp. | 1 | 8月 | A | |
| Xylobates属sp. | 1 | 8月 | A | |
| Podocinum属sp. | 2 | 8月 | A | |
| トゲダニ目spp. | 4 | 8月 | A | |
| Pheretima属sp. | 1 | 8月 | A | |
| シマツノアヤトビムシ | 1 | 8月 | A | |
| アイイロハゴロモトビムシ | 12 | 8月 | A | |
| シロフォルソムトビムシ | 22 | 8月 | A | |
| コガタドウナガツチトビムシ | 2 | 8月 | A | |
| Pseudachorutes属 | 2 | 8月 | A | |
| ニセハリアリ | 4 | 8月 | A | |
| アメイロアリ | 4 | 8月 | A | |
| キタウロコアリ | 3 | 8月 | A | |
| ヒラタウロコアリ | 3 | 8月 | A | |
| コツノアリ | 4 | 8月 | A | |
| ホシカメムシ科sp.幼虫 | 1 | 8月 | A | |
| ナミツブダニ | 1 | 8月 | A | |
| コムカデ綱 | 3 | 8月 | B | |
| キリフリヒトフシムカデ | ♀ | 1 | 8月 | B |
| ワタナベヒトフシムカデ酷似種 | ♂ | 1 | 8月 | B |
| ナスヒトフシムカデ | ♂ | 1 | 8月 | B |
| Monotarsobius属sp. | 3 | 8月 | B | |
| オカダンゴムシ | 1 | 8月 | B | |
| Xylobates属sp. | 1 | 8月 | B | |
| フトツツハラダニ | 10 | 8月 | B | |
| トウキョウフリソデダニ | 1 | 8月 | B | |
| Holaspina属sp. | 3 | 8月 | B | |
| Podocinum属sp. | 1 | 8月 | B | |
| トゲダニ目spp. | 5 | 8月 | B | |
| ニホンケシガイ | 1 | 8月 | B | |
| カマアシムシ目sp. | 1 | 8月 | B | |
| シマツノアヤトビムシ | 1 | 8月 | B | |
| ヒメフォルソムトビムシ | 49 | 8月 | B | |
| アイイロハゴロモトビムシ | 2 | 8月 | B | |
| フタツメフォルソムトビムシ | 2 | 8月 | B | |
| ヨシイフォルソムトビムシ | 5 | 8月 | B | |
| ヒメトゲトビムシ | 2 | 8月 | B | |
| フクロムラサキトビムシ | 1 | 8月 | B | |
| キタウロコアリ | 1 | 8月 | B | |
| ニセハリアリ | 1 | 8月 | B | |
| ウメマツアリ | 1 | 8月 | B | |
| コツノアリ | 69 | 8月 | B | |
| イガウロコアリ | 1 | 8月 | B | |
| コメツキムシ科sp. | 1 | 8月 | B | |
| フトツツハラダニ | 1 | 8月 | B | |
| ヤブコウジハカマカイガラムシ. | 3 | 8月 | C | |
| コムカデ綱 | 5 | 8月 | C | |
| Monotarsobius属sp. | ♂ | 4 | 8月 | C |
| Mecistocephalus属sp. | 2 | 8月 | C | |
| ヒロズジムカデ | 3 | 8月 | C | |
| フイリワラジムシ | 6 | 8月 | C | |
| イトダニ科sp. | 6 | 8月 | C | |
| Holostaspella属sp. | 4 | 8月 | C | |
| トゲダニ目spp. | 6 | 8月 | C | |
| Xylobates属sp. | 8 | 8月 | C | |
| ハナビラオニダニ | 1 | 8月 | C | |
| アジアオニダニ | 6 | 8月 | C | |
| ヒメイブリダニ | 1 | 8月 | C | |
| カマアシムシ目sp. | 8 | 8月 | C | |
| コガタドウナガツチトビムシ | 13 | 8月 | C | |
| シマヤマトビムシ | 2 | 8月 | C | |
| アイイロハゴロモトビムシ | 2 | 8月 | C | |
| フクロムラサキトビムシ | 2 | 8月 | C | |
| キイロシリアゲアリ | 1 | 8月 | C | |
| コツノアリ | 5 | 8月 | C | |
| キタウロコアリ | 2 | 8月 | C | |
| コナカイガラムシ科sp. | 2 | 8月 | D | |
| コツノアリ | 1 | 8月 | D | |
| ミコシヤスデ科sp. | 1 | 8月 | D | |
| オウギヤスデ | 3 | 8月 | D | |
| ハガヤスデ属 | 2 | 8月 | D | |
| ヒロズジムカデ | 2 | 8月 | D | |
| Monotarsobius属sp. | 1 | 8月 | D | |
| フイリワラジムシ | 9 | 8月 | D | |
| Xylobates属sp. | 1 | 8月 | D | |
| ヤマトクモスケダニ | 4 | 8月 | D | |
| Lasioseius属sp. | 3 | 8月 | D | |
| Cybaeus属sp. | 2 | 8月 | D | |
| アイイロハゴロモトビムシ | 19 | 8月 | D | |
| ヨシイフォルソムトビムシ | 2 | 8月 | D | |
| ヒメフォルソムトビムシ | 6 | 8月 | D | |
| コガタドウナガトビムシ | 2 | 8月 | D | |
| Pseudachorutes属sp. | 4 | 8月 | D | |
| カマアシムシ目sp. | 1 | 8月 | D | |
| ヤマトシロアリ | 19 | 8月 | D | |
| コツノアリ | 5 | 8月 | D | |
| コメツキムシ科sp.幼虫 | 1 | 8月 | D |
Rで群集構造解析を行うには?
以上のデータから『R』で群集構造解析を行うことができます。以下がその全てのコードです。今回は『R 4.3.2』を使用しています。基本的な内容は高田(2018)を参照しました。土居・岡村(2011)もご参照ください。
required_packages <- c("here", "vegan", "labdsv")
# パッケージがインストールされていなければインストールする
installed_packages <- rownames(installed.packages())
for (pkg in required_packages) {
if (!pkg %in% installed_packages) {
install.packages(pkg)
}
}
# パッケージをロードする
lapply(required_packages, library, character.only = TRUE)
#ディレクトリの指定
getwd()
script_directory <- here("./土壌動物の室内分析と群集構造解析の例")
setwd(script_directory)
#分析データの読み込み
soil_data <- read.csv("土壌動物の個体数データ.csv", header = TRUE, row.names = 1, fileEncoding = "UTF-8")
# 生物多様性の計算
shannon_diversity <- diversity(soil_data, index = "shannon")
shannon_diversity
simpson_diversity <- diversity(soil_data, index = "simpson")
simpson_diversity
renyi(soil_data)
renyi_diversity <- exp(renyi(soil_data))
print(renyi_diversity)
# メタデータの多次元尺度構成法(MDS)解析
soil_data_matrix <- data.matrix(soil_data) # 行列に変換
mds_result <- metaMDS(soil_data_matrix, dist = "bray", autotransform = FALSE)
mds_result
ordipointlabel(mds_result, display = "sites", cex = 1, col = "black", pch = 16)
# クラスター解析
veg_dist <- vegdist(soil_data_matrix, method = "bray")
veg_dist
hierarchical_clustering <- hclust(veg_dist, method = "ward.D2")
hierarchical_clustering
plot(hierarchical_clustering, hang = -1)
# クラスタ数の評価
average_silhouette <- numeric(nrow(soil_data))
for (i in 2:(nrow(soil_data) - 1)) {
silhouette_vals <- silhouette(cutree(hierarchical_clustering, k = i), veg_dist)
average_silhouette[i] <- mean(silhouette_vals[, 3])
}
average_silhouette
# クラスタリングの結果
optimal_cluster <- cutree(hierarchical_clustering, k = which.max(average_silhouette))
# Silhouetteプロット
silhouette_vals <- silhouette(optimal_cluster, veg_dist)
rownames(silhouette_vals) <- row.names(soil_data)
plot(silhouette_vals)
# PAM法によるクラスタリングと重要な生物種の特定
pam_result <- pam(veg_dist, k = which.max(average_silhouette), diss = TRUE)
ind_val_analysis <- indval(soil_data, pam_result$clustering, numitr = 5000)
significant_groups <- ind_val_analysis$maxcls[ind_val_analysis$pval <= 0.05]
significant_species <- ind_val_analysis$indcls[ind_val_analysis$pval <= 0.05]
p_values <- ind_val_analysis$pval[ind_val_analysis$pval <= 0.05]
frequencies <- apply(soil_data > 0, 2, sum)[ind_val_analysis$pval <= 0.05]
significant_results <- data.frame(group = significant_groups, indval = significant_species, pvalue = p_values, freq = frequencies)
significant_results_summary <- significant_results[order(significant_results$group, -significant_results$indval), ]
significant_results_summary
データファイルの読み込み方は?
最初に今回使用するパッケージをインストールしておきます。既にインストールしている人は不要です。
required_packages <- c("here", "labdsv", "vegan")
install.packages(required_packages)
install.packages(required_packages)の代わりに以下のコードを使用すると既にインストールしているパッケージはスキップします。
installed_packages <- rownames(installed.packages())
for (pkg in required_packages) {
if (!pkg %in% installed_packages) {
install.packages(pkg)
}
}
慣れないうちは個別に読み込んでも良いですが、上のコードの方がロードのときにベクトルにしたパッケージ名を使い回せます。
install.packages(here)
install.packages(labdsv)
install.packages(vegan)
そして、パッケージをロードしましょう。
lapply(required_packages, library, character.only = TRUE)
同様に慣れないうちは個別に読み込んでも構いません。
library(here)
library(labdsv)
library(vegan)
次にディレクトリを指定します。ディレクトリは絶対パスでも指定は可能ですが、より簡単な現在使用しているワークスペースを作業ディレクトリに指定してくれるhereパッケージのhere()関数を使用しています。here()関数を使用するとGoogleドライブなどのクラウドストレージでファイル共有したり、別PCに以降したときでもパスが有効なので便利です。
ただ、注意したいのがhere()関数はワークスペースのパスを呼び出す関数なので、環境によっては(私の場合はVS code)、開いている.Rファイルの上のパスまでは指定してくれないので、ここでは1つ上のパス名を手入力しています。この点はなにか改善点が欲しいところです。
getwd()
script_directory <- here("./土壌動物の室内分析と群集構造解析の例")
setwd(script_directory)
そして、.csvファイルを読み込みます。注意したいのが.csvファイルの文字コードはUTF-8でなければなりません。この仕様は最近のバージョンで追加されたようで古いRコードで実行しようとした時に困りました。(これはもしかしたら私がVS codeで実行しているせいかもしれません。)
OSがWindowsのPCでは『Microsoft Excel(エクセル)』などで.xlsxファイルから.csvファイルを作成すると、文字コードがShift-JIS(ANSI)の.csvファイルが作成されてしまいます。
そのため、「名前をつけて保存」をするときに「CSV」ではなく、「CSV UTF-8」と書いてある方を選択しましょう。これは本当に勘違いしやすいです。
一応、read.csv()関数の引数を「fileEncoding = "Shift-JIS"」とすることでShift-JISでも読み込めることになっていますが、そもそもShift-JISが古い規格で現在ではUTF-8が上位互換の文字コードであり、そのままの.csvファイルを使用するのはおすすめできません。
以下では念の為「fileEncoding = "UTF-8"」で指定しています。
soil_data <- read.csv("土壌動物の個体数データ.csv", header = TRUE, row.names = 1, fileEncoding = "UTF-8")
以上でデータの読み込みは完了です。
なお、.csvファイルの読み込みでよくある失敗は列と行の向きとラベルの有無です。
実際に今回読み込んでいる.csvファイルは上記の表の列と行が反対になったものを読み込んでいます(「性別」の行も削除しています)。『Microsoft Excel』を使用している場合は、右クリックして「貼り付けのオプション」から「行/列の入れ替え」を選択することで簡単に反転させられます。
また列と行どちらにもラベルがある場合は、「header = TRUE, row.names = 1」と指定しましょう。
Rを用いてシャノンの多様度指数とシンプソンの多様度指数を計算するには?
まずは複数ある生物多様度指数を計算してみましょう。
シャノンの多様度指数(Shannon index)を計算し、内容を確認するには以下のコードを使用します。
shannon_diversity <- diversity(soil_data, index = "shannon")
shannon_diversity
今回は以下の数値が得られました。
| A(5月) | B(5月) | C(5月) | D(5月) | A(8月) | B(8月) | C(8月) | D(8月) |
| 3.081413 | 2.722761 | 2.739502 | 2.701395 | 2.820675 | 1.958188 | 2.829526 | 2.496044 |
シンプソンの多様度指数(Simpson index)を計算し、内容を確認するには以下のコードを使用します。
simpson_diversity <- diversity(soil_data, index = "simpson")
simpson_diversity
今回は以下の数値が得られました。
| A(5月) | B(5月) | C(5月) | D(5月) | A(8月) | B(8月) | C(8月) | D(8月) |
| 0.9319903 | 0.9061634 | 0.8824365 | 0.8974433 | 0.8955017 | 0.7420609 | 0.9303704 | 0.8756084 |
いずれの多様度指数でもA(5月)が最も多様性が高く、B(8月)が低い地点となっています。一般に8月の方が昆虫も増え、多くなるようにも思えますが、意外な結果となりました。
Rを用いてNMDSをプロットするには?
次に多変量解析の一種である非計量多次元尺度法(Nonmetric Multidimensional Scaling、NMDS)を行っていきましょう。NMDSはデータセット内の個々のデータポイント間の類似性または距離を反映するように、データを低次元の空間に配置する手法です。
要するに今回の場合は図の中でプロットが近いほど生物群集構造が近いということを意味します。
Rで非計量多次元尺度法をプロットするには以下のコードを使用します。
soil_data_matrix <- data.matrix(soil_data) # 行列に変換
mds_result <- metaMDS(soil_data_matrix, dist = "bray", autotransform = FALSE)
mds_result
ordipointlabel(mds_result, display = "sites", cex = 1, col = "black", pch = 16)
その結果、以下の図が得られました。

NMDSからは季節による土壌動物群集の影響が考えられます。一般に土壌動物は季節の影響を受けにくいとされていますが、今回は夏季に昆虫類が増加したことが影響したと思われます。また地点の影響も出ていますが、D地点では大きく異なる結果になっています。D地点は今回のデータでは地点の環境に関する情報がありませんが、季節で大きく環境がかわる地点であった可能性もあります。なお、環境に関する量的データがあれば更にその影響を評価したものをプロットできます。
Rを用いてクラスター解析を行うには?
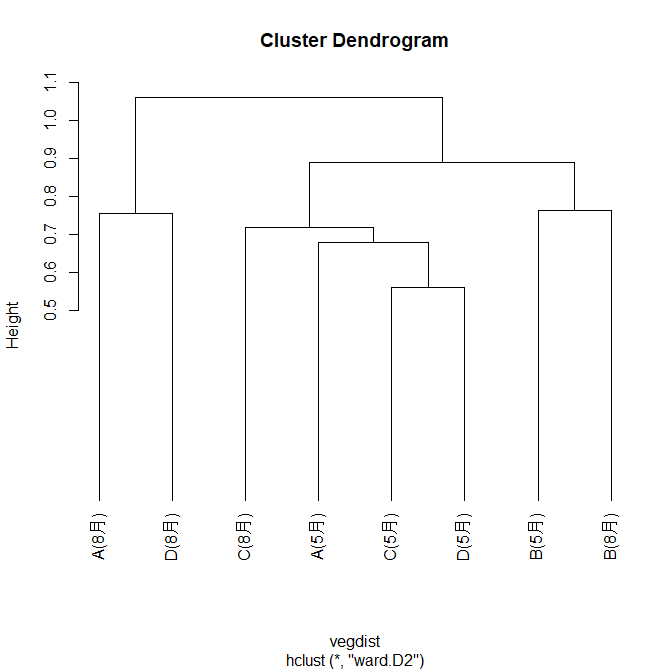
次にクラスター解析です。クラスター解析はデータセット内の個々のデータ点を似た特性や属性に基づいてグループにまとめる統計的手法で、データ内のパターンや構造を把握し、データを理解することができます。今回は地点間で群集構造が近い順にまとめることになります。クラスター解析とプロットは以下のコードで行えます。
今回の解析にはブレイ・カーチス非類似度(Bray-Curtis dissimilarity)を使用しています。
veg_dist <- vegdist(soil_data_matrix, method = "bray")
veg_dist
hierarchical_clustering <- hclust(veg_dist, method = "ward.D2")
hierarchical_clustering
plot(hierarchical_clustering, hang = -1)
その結果、以下の図が得られました。
これを以下のコードでシルエット分析(Silhouette Analysis)を行います。クラスタリングの結果を評価し、各データポイントがその所属するクラスター内でどれだけ密集しているかを測定する手法です。シルエット分析はクラスタリングの妥当性を評価するための一つの手法であり、クラスターの数や形状を決定する際に有用です。
要するに恣意的になりがちなグループ分割を避けて、意味がありそうなグループの分割基準を調べてくれる便利なものです。
silhouette_vals <- silhouette(optimal_cluster, veg_dist)
rownames(silhouette_vals) <- row.names(soil_data)
plot(silhouette_vals)
その結果、以下の図が得られ、2つ(A(8月) とD(8月)のクラスターとその他のクラスター)に分割することが最適と判断されました。
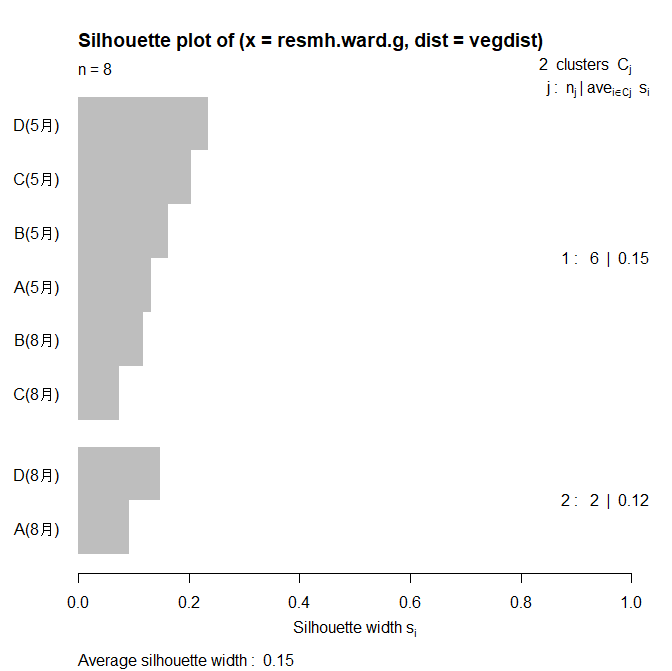
cutree()関数を使用すると改めて各地点が具体的にどのグループに属するかを調べることができます。
| A(5月) | B(5月) | C(5月) | D(5月) | A(8月) | B(8月) | C(8月) | D(8月) |
| 1 | 1 | 1 | 1 | 2 | 1 | 1 | 2 |
クラスター解析では今回の分析例では土壌動物の種ごとの生態に関する情報が極めて少なく、また地点の植生など環境情報が不足しているので考察は難しいですが、これらの情報も加味すればより深い考察が可能です。
Rを用いて指標種分析を行うには?
最後にクラスター解析に基づいた指標種分析を行います。以下のコードで行えます。PAM法を使用して、最適なクラスタ数を決定し、データをクラスタリングし、クラスタリング結果を元に、各クラスタごとの重要な生物種を特定します。 そして、特定された重要な生物種に関する情報(クラスター・種・p値・出現頻度)を収集し、クラスタごとに重要な生物種をまとめ、結果を要約しています。
pam_result <- pam(veg_dist, k = which.max(average_silhouette), diss = TRUE)
ind_val_analysis <- indval(soil_data, pam_result$clustering, numitr = 5000)
significant_groups <- ind_val_analysis$maxcls[ind_val_analysis$pval <= 0.05]
significant_species <- ind_val_analysis$indcls[ind_val_analysis$pval <= 0.05]
p_values <- ind_val_analysis$pval[ind_val_analysis$pval <= 0.05]
frequencies <- apply(soil_data > 0, 2, sum)[ind_val_analysis$pval <= 0.05]
significant_results <- data.frame(group = significant_groups, indval = significant_species, pvalue = p_values, freq = frequencies)
significant_results_summary <- significant_results[order(significant_results$group, -significant_results$indval), ]
significant_results_summary
このコードを実行すると以下の表が得られます。
| group | indval | pvalue | freq | |
| フクロムラサキトビムシ | 1 | 1 | 0.0378 | 6 |
| Pseudachorutes属sp. | 2 | 1 | 0.0374 | 2 |
これにより、A(8月) とD(8月)のクラスターはヤマトビムシ属(Pseudachorutes)で指標され、その他のクラスターはフクロムラサキトビムシで指標されることがわかりました。残念ながらこれらの種についても生態に関する情報が不足していますが、よく生態が知られている種で分析を行えば、より深い考察が可能です。
これらの解析は昆虫・脊椎動物・植物・水生生物・細菌など様々な生物群集でも可能です。
引用文献
土居秀幸・岡村寛. 2011. 生物群集解析のための類似度とその応用:Rを使った類似度の算出、グラフ化、検定. 日本生態学会誌 61(1): 3-20. https://doi.org/10.18960/seitai.61.1_3
高田宜武. 2018. 水産研究・教育機構「日本海区水産研究所」:Rによる群集組成の解析. https://jsnfri.fra.affrc.go.jp/gunshu/index.html


